# 2. Upload deltaG_values.csv of every docking to generate a boxplot to obtain a summary table transposed data in csv and boxplot
# Has de pujar els diferents arxius i penjar cancel quan acabis, posar els noms en català (en aquest exemple: Taxinina A, etc)
# Després has d'escriure el codi PDB de la teva proteïna (en aquest exemple 4HFZ)
!pip install pandas matplotlib io numpy
#Aqui instalem les biblioteques python, pandas ens permet treballar amb csv facilment, matplotlib ens peermet dibuixar grafics, io ens permet entrades (imput) i sortides
from google.colab import files
import pandas as pd
import matplotlib.pyplot as plt
import io
import numpy as np
# Initialize an empty list to store DataFrame objects
dfs = []
# Upload CSV files one by one
print("Upload CSV files one by one. Press Cancel to stop uploading.")
while True:
uploaded_files = files.upload()
if len(uploaded_files) == 0:
break
for filename, contents in uploaded_files.items():
# Read CSV file as DataFrame and append it to the list
df = pd.read_csv(io.StringIO(contents.decode('utf-8')), header=None)
# Add a column to identify the compound
df['Compound'] = f'Compound {chr(ord("A") + len(dfs))}'
dfs.append(df)
# Concatenate DataFrames vertically
combined_df = pd.concat(dfs, ignore_index=True)
# Transpose the DataFrame so that rows become columns
transposed_df = combined_df.set_index('Compound').T
# Save the transposed DataFrame to a new CSV file
transposed_csv_path = 'transposed_data.csv'
transposed_df.to_csv(transposed_csv_path)
# Prompt the user to enter real chemical names for each compound
real_names_mapping = {}
for i, df_name in enumerate(transposed_df.columns):
real_name = input(f"Enter the real chemical name for {df_name}: ")
real_names_mapping[df_name] = real_name
# Prompt the user to enter the last word of the graph title
graph_title_suffix = input("Enter the last word of the graph title: ").strip()
# Create a customized boxplot for compounds
plt.figure(figsize=(8, 6))
# Set colors
box_color = 'blue'
median_color = 'orange'
whisker_color = 'green'
cap_color = 'purple'
# Create a boxplot
boxprops = dict(color=box_color)
medianprops = dict(color=median_color)
whiskerprops = dict(color=whisker_color)
capprops = dict(color=cap_color)
boxplot = transposed_df.boxplot(vert=False, return_type='dict', boxprops=boxprops, medianprops=medianprops, whiskerprops=whiskerprops, capprops=capprops)
# Overlay individual data points
for df_name in transposed_df.columns:
y = np.random.normal(list(transposed_df.columns).index(df_name) + 1, 0.1, size=len(transposed_df[df_name]))
plt.scatter(transposed_df[df_name], y, alpha=0.5, s=10)
# Set ticks and labels
plt.yticks(np.arange(1, len(transposed_df.columns) + 1), [real_names_mapping[col] for col in transposed_df.columns])
plt.xlabel("Energia d'unió (kcal/mol)")
plt.ylabel("Lligands")
plt.title(f"Acoblament molecular amb proteïna PDB {graph_title_suffix}")
plt.grid(True)
plt.axvline(x=0, color='red', linestyle='--') # Add line at 0 for reference
plt.tight_layout()
# Save the plot as an image file
plot_image_path = 'boxplot.png'
plt.savefig(plot_image_path)
# Download the transposed CSV file and the plot image
files.download(transposed_csv_path)
files.download(plot_image_path)
# Print paths to the saved files
print("Transposed data saved to:", transposed_csv_path)
print("Plot image saved to:", plot_image_path)
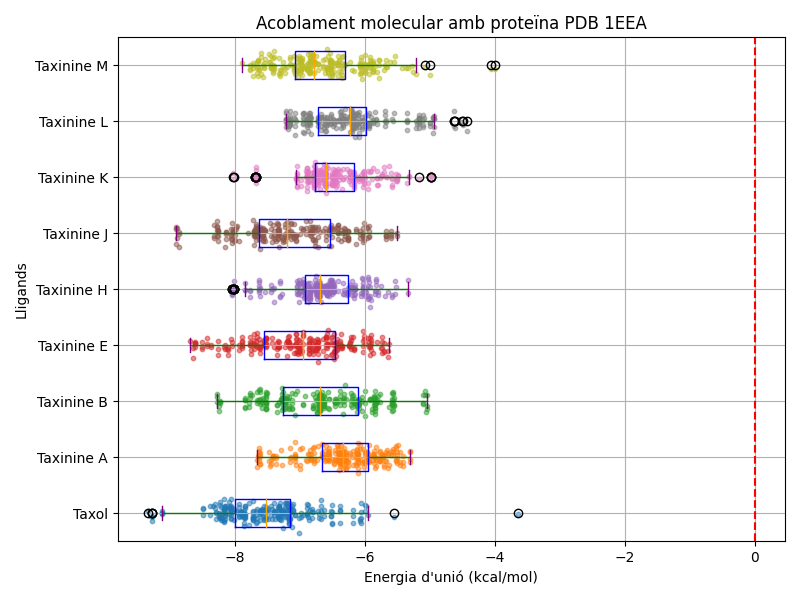
Taxinines i Taxol contra el càncer
Aquí es poden veure els gràfics sobre quin compost és més actiu per la proteïna 1FOS, el resultat que he obtingut és que el Taxol és el més actiu.
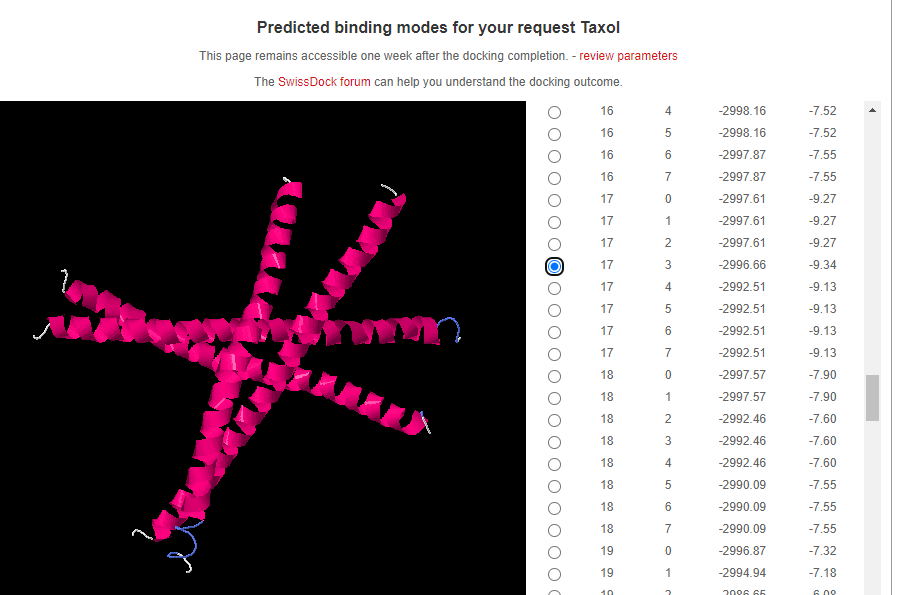
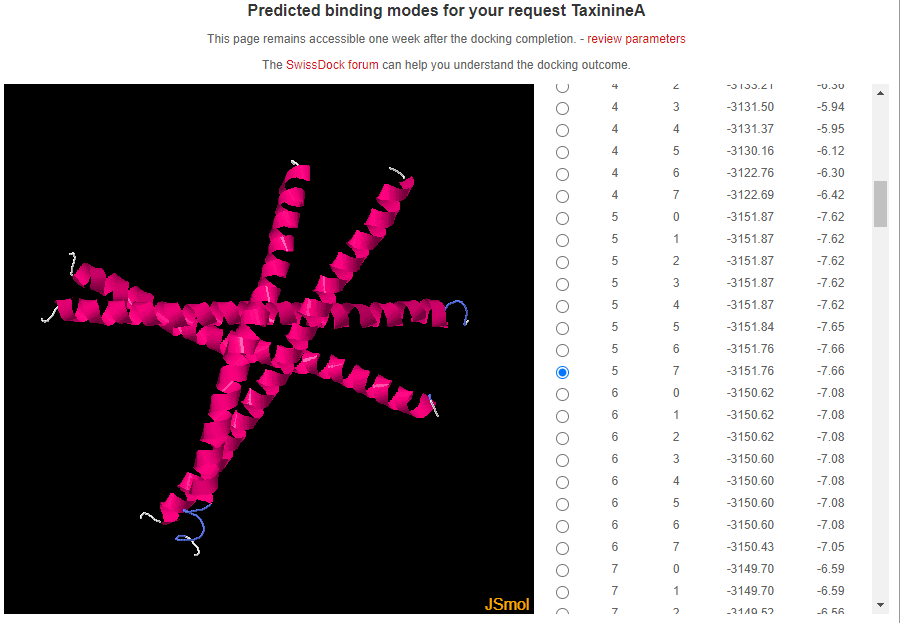
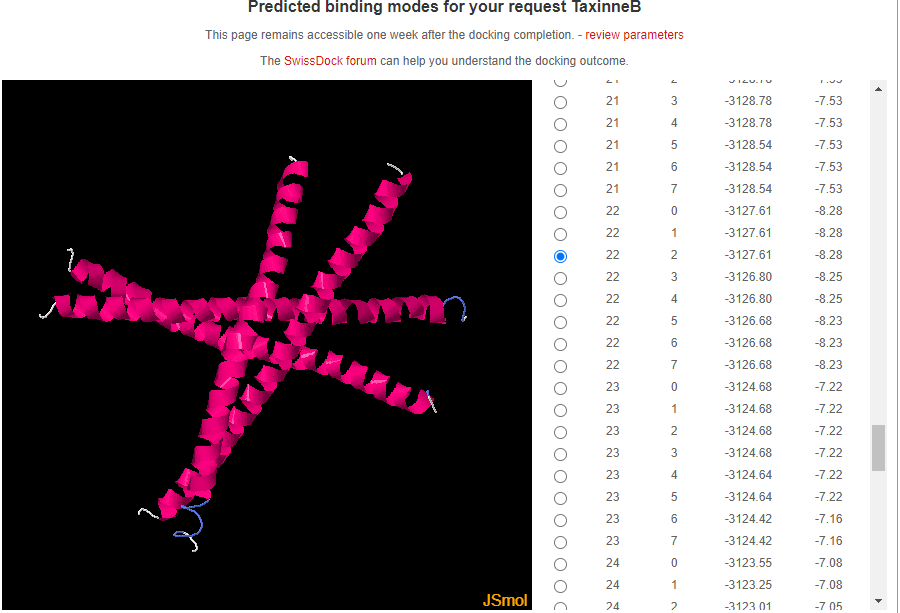
En conclusió, el taxol mostra una activitat més elevada que la taxinina en la proteïna 1FOS. Aquesta major activitat es pot atribuir a la seva major afinitat d'unió i estabilització de la proteïna, aquest fet ens fa veure el potencial superior del taxol en aplicacions mèdiques relacionades amb la proteïna 1FOS.